Yasemin Şaplakoğlu
Çev. Cem Oran
Okuyacağınız makale, quantamagazine.org’da 26 Haziran 2024’de yayınlandı. Yazar Yasemin Saplakoglu, Quanta Magazine’in biyoloji alanında popüler bilim içeriği üreten kadrolu yazarı. Connecticut Üniversitesi’nden biyomedikal mühendisliği, California Üniversitesi, Santa Cruz’da ise bilim iletişimi alanında yüksek lisans derecelerine sahip. Arkadaşımız Cem Oran bu ilginç makaleyi Bilim ve Gelecek okurları için Türkçeleştirdi.
Aralık 2020’de, pandemi kısıtlamalarının yüz yüze toplantıları imkânsız hale getirdiği bir dönemde, yüzlerce hesaplamalı bilimci ekranlarının önünde toplanarak bilimde yeni bir çağın açılışını izlediler.
Bir konferans için toplanmışlardı. Bazılarının neredeyse otuz yıldır kişisel olarak katıldığı bu konferans aynı zamanda hem dostane bir yarışma hem de hepsinin bir araya gelip aynı soruna kitlendikleri bir etkinlikti. Protein katlanması problemi olarak bilinen bu sorun basit bir şekilde ifade edilebiliyordu: Bir protein molekülünün üç boyutlu şekli, onun en temel bilgisi olan bir boyutlu moleküler kodundan doğru bir şekilde tahmin edilebilir mi? Proteinler, hücrelerimizi ve vücudumuzu canlı ve çalışır durumda tutar. Bir proteinin şekli onun davranışını belirlediğinden bu problemi başarılı bir şekilde çözmek, hastalıkların anlaşılmasından yeni ilaçların üretimine ve yaşamın işleyişine dair derinlemesine bir kavrayış edinilmesine kadar geniş bir etki yaratabilir.
Her iki yılda bir düzenlenen konferansta, biliminsanları en yeni protein katlanması araçlarını test ediyorlardı. Ancak çözüm her defasında erişilemezliğini sürdürmüştü. Bazıları, bu tahminlerde sadece kısmi bir gelişme sağlamak için tüm kariyerlerini harcamışlardı. Bu yarışmalar, küçük adımlarla ilerleyerek sürmüştü ve araştırmacıların 2020’nin farklı olacağını düşünmek için pek bir nedeni yoktu. Ama bu konuda yanılmışlardı.
O hafta, protein bilimi topluluğuna nispeten yeni bir katılımcı olan John Jumper, Google DeepMind’ın Londra’daki yapay zekâ kolundan çıkan yeni bir yapay zekâ aracı olan AlphaFold2’yi tanıttı. Zoom üzerinden sunduğu veriler, AlphaFold2’nin üç boyutlu protein yapılarındaki tahmin modellerinin % 90’ın üzerinde doğruluk sunduğunu gösterdi. Bu sonuç, en yakın rakibinden beş kat daha iyi sonuçlar verdiği anlamına geliyordu.
O an itibariyle, protein katlanması probleminin çözümü imkânsız olmaktan çıktı. İnsan zekâsının başarısız olduğu yerde yapay zekânın başarıya ulaşması biyologlar topluluğunu sarsmıştı. Toplantıya katılan Columbia Üniversitesi Matematiksel Genomik Programı’ndan sistem biyoloğu Mohammed AlQuraishi, “Şoktaydım” diyor. “Birçok kişi inkâr içindeydi.”
Ancak konferansın kapanış konuşmasını yaparken organizatör John Moult şüpheye yer bırakmadı: AlphaFold2 “büyük ölçüde” protein katlanması sorununu çözmüştü ve protein bilimini sonsuza dek değiştirmişti. Siyah bir kazak giymiş olarak ev ofisindeki kitaplığının önünde oturan Moult, Zoom üzerinden slaytlarına tıklarken heyecanlı ama aynı zamanda karanlık bir tonda konuştu: “Bu bir son değil, bir başlangıçtır.”
Google’ın halkla ilişkiler makinesi bu haberi dünyaya yaydığında, bilim medyası çılgına döndü. Başlıklar, AlphaFold2’nin “her şeyi değiştireceğini” iddia etti. Tek bir proteinin yapısını inceleyerek bütün kariyerlerini geçirmiş protein biyologlarını işlerini kaybetme korkusu sardı. Bazıları AlphaFold2’nin ilaç geliştirme süreçlerini devrim niteliğinde değiştireceğini öne sürdü. Biyologlar artık proteinlerin şekillerini hızlıca öğrenebileceğinden dolayı bu proteinleri hedefleyen yeni ilaçlar üretebileceklerdi. Diğerleri ise sonuçların çoğunlukla abartı olduğunu ve pek bir şeyin değişmeyeceğini savundu.
Moult’un kendisi bile bu gelişmeyi tam olarak kavrayamamıştı. Konferansı herkesin aklındaki soru ile sonlandırdı: “Peki şimdi ne olacak?”
Bu üç buçuk yıl önceydi. Bugün nihayet bu soruya cevap verebilmeye başlıyoruz. AlphaFold2, biyologların proteinleri inceleme sistematiğini şüphesiz değiştirdi. Ancak AlphaFold2 güçlü bir tahmin aracı olmasına rağmen, her şeyi bilen bir makine değil. Protein katlanması probleminin bir kısmını çok pürüzsüzce çözdü, ama bir biliminsanı gibi değil. Biyolojik deneylerin yerini almadı; aksine onlara olan ihtiyacı vurguladı.
Belki de AlphaFold2’nin en büyük etkisi, biyologların yapay zekânın gücüne dikkatini çekmesi olmuştur. Doğada bulunmayan yeni proteinleri tasarlayan algoritmalar da dahil olmak üzere yeni algoritmalara; yeni biyoteknoloji şirketlerine ve bilim pratiği için yeni yöntemlere ilham verdi. Mayıs 2024’te duyurulan halefi AlphaFold3, DNA veya RNA gibi diğer moleküllerle kombinasyon içindeki proteinlerin yapılarını modellemeyi başararak biyolojik tahminin bir sonraki aşamasına geçti.

AlQuraishi, “Bu, en büyük ‘bilimde makine öğrenmesi’ hikayesidir” diyor. Ancak yapay zekânın henüz doldurmadığı büyük boşluklar var. Bu araçlar, proteinlerin zaman içinde nasıl değiştiğini simüle edemez veya onları hücre içindeki bağlamında modelleyemezler. Lawrence Berkeley Ulusal Laboratuvarı’nda biyomoleküllerin yapılarını modelleyen algoritmalar geliştiren yapı biyoloğu Paul Adams, “AlphaFold her şeyi değiştirdi ve hiçbir şeyi değiştirmedi” diyor.
Bu yazı, Jumper’ın Google DeepMind’daki ekibinin protein bilimindeki hamlesini nasıl gerçekleştirdiğinin ve biyolojide yapay zekânın geleceği için ne anlama geldiğinin hikayesidir.
SORUNUN FORMÜLE EDİLMESİ
Bir origami kağıdının, belirli şekillerde katlanana kadar basılmış odun hamurundan pek bir farkı yoktur; ancak sonrasında yeni bir şey halini alır. Birkaç hassas katlama ve döndürme hareketiyle, kâğıt bir falcıya dönüşür, geleceğinizi tahmin eden kâğıttan bir cihaza. Aynı kâğıdı alıp bazı katlama adımlarını değiştirirseniz, şimdi kanatlı bir turna halini alır ve sahibine iyi şans getirir.
Benzer şekilde, uzun bir aminoasit molekülü dizisi, kendiliğinden doğal şeklini alana kadar işlevsizdir. Biyologların yapı olarak adlandırdığı bu şekil, proteinin nasıl diğer moleküllerle bağlandığını veya onlarla etkileşime girdiğini belirler ve dolayısıyla bir hücredeki rolünü tanımlar.

Gezegenimizde birkaç yüz milyon bilinen protein var ve çok daha fazlası bilinmiyor. Proteinler hemen her şeyi yaparlar: Hemoglobin ve miyoglobin, oksijeni kaslar ve vücut boyunca taşır. Keratin, saç, tırnak ve cilde yapı verir. İnsülin, glukozun hücrelere hareket etmesini ve enerjiye dönüştürülmesini sağlar. Proteinler, hayatlarında yaptıkları sayısız işe uyacak şekilde neredeyse sonsuz sayıda şekil alabilirler. AlQuraishi, “Atomlardan ekosistemlere kadar, [protein yapısı] bir tür ortak dil gibidir” diyor. “Onlar her şeyin gerçekleştiği yerdir.”
Bir hücre, aminoasitler adı verilen küçük molekülleri uzun polipeptit dizileri halinde zincirleme bağlayarak proteinleri üretir. Seçtiği aminoasitler, DNA tarafından sağlanan temel talimat setine bağlıdır. Bir polipeptid zinciri yaratılmasıyla birlikte çok kısa bir süre içerisinde eğilir, burkulur, katlanır ve proteinin nihai üç boyutlu şeklini alır. Bu moleküler montaj hattından çıkan proteinler biyolojik işini yapmak üzere harekete geçerler.
Eğer proteinler bu katlanma sürecini mükemmel bir şekilde gerçekleştiremeseydi, vücutta felaketler zinciri meydana gelirdi. Yanlış katlanmış veya çözülmüş bir protein, toksisiteye ve hücre ölümüne yol açabilir. Orak hücre anemisi gibi birçok hastalık ve bozukluk, yanlış katlanmış proteinlerden kaynaklanır. Yanlış katlanmış proteinler, aynı zamanda, Alzheimer ve Parkinson gibi nörodejeneratif hastalıkların ayırt edici özellikleri olan kümeler halinde bir araya gelebilir. Ancak, protein katlanmasının tam olarak nasıl gerçekleştiğini kimse bilmiyor. Bu basit moleküler zincirlerdeki dizilim bilgisi, bir proteinin karmaşık şeklini nasıl kodlar? Bu, “sorabileceğimiz en derin sorudur” diyor Johns Hopkins Üniversitesi’nden emekli biyofizik profesörü George Rose.
Biliminsanları bu soruyu 1930’lardan beri araştırıyorlardı. Ancak çalışmalar, 1950’lerin ortalarında biyokimyacı Christian Anfinsen’ın proteinleri kimyasal çözeltilere eklediğinde gerçek bir sıçrama yaptı. Bu çözeltiler, proteinleri bağlarını kırarak açıyor veya yanlış katlanmalarına neden oluyordu. Çözeltiye eklediği proteinlerin daha sonrasında neler yaptıklarını gözlemleyen Anfinsen, açılmış veya yanlış katlanmış proteinlerin kendiliğinden doğru yapılarına yeniden döndüklerini gördü. İleride ona Nobel Ödülü kazandıracak olan bu bulgular, proteinlerin üç boyutlu şekillerini -aminoasit dizilerinin yazdığı- içsel bir kod ile oluşturduklarını gösterdi. Anfinsen, buradan yola çıkarak bir proteinin şeklinin aminoasit dizisinden tahmin edilebileceğini öne sürdü. Bu, genel olarak protein katlanması problemi olarak tanınır oldu.
Bir protein, polipeptid zinciri bir araya geldikten sonra, saniyenin binde biri gibi kısa bir sürede nihai yapısına katlanabilir. 1969 tarihli “Nasıl Zarifçe Katlanılır” (How to Fold Graciously) başlıklı makalesinde moleküler biyolog Cyrus Levinthal, bir proteinin her olası katlanma seçeneğini denemesi durumunda, montajının imkânsız derecede uzun süreceğini hesapladı. Levinthal bir şeyin proteini doğrudan doğruya katlanma yoluna yönlendirmesi gerektiğini düşündü.
Zamanla, protein katlanması problemi farklı türde sorulara ayrıldı. Şu an için üç ana soru göze çarpıyor: Bir proteinin yapısı aminoasit dizisinden tahmin edilebilir mi? Katlanma kodu nedir? Ve katlanma mekanizması nedir?
Bu sorular, deneysel olarak belirlenen ilk protein yapılarının ortaya konulduğu 1960’ların başlarında biliminsanlarının zihinlerine girmeye başladı. Cambridge Üniversitesi’nden biyologlar Max Perutz ve John Kendrew, proteinleri kristallere büyüttü, onları X-ışınlarıyla bombardıman etti ve ışınların nasıl büküldüğünü ölçtü (bu teknik X-ışını kristalografisi olarak bilinir). Bu şekilde hemoglobin ve miyoglobin’in üç boyutlu yapılarını belirlediler. Bu işlem yirmi yıldan fazla sürdü ve bu keşifleri için Nobel Ödülü’nü paylaştılar.
O zamandan beri, sayısız araştırmacı, sadece farklı proteinlerin neye benzediğini değil, nasıl bu şekilde göründüklerini anlamaya çalıştı. Glasgow Üniversitesi’nden yapı biyoloğu Helen Walden, “Bir şeylerin nasıl göründüğünü bilmek istemek çok insani bir şeydir, çünkü o zaman nasıl işlediğini anlayabilirsiniz” diyor. Bazıları problemin kimyasını, diğerleri fiziğini araştırdı. Deneyciler, protein şekillerini titiz laboratuvar çalışmalarıyla yeniden inşa ettiler. Hesaplamalı biyologlar, modeller ve simülasyonlarla ipuçları aradılar, bu modelleri ve simülasyonları çeşitli algoritmik kurallar kombinasyonlarıyla tekrar tekrar programladılar.
Daha fazla yapı elde edildikçe, protein bilimi topluluğu bunları düzenleyip paylaşmanın bir yoluna ihtiyaç duydu. 1971’de Protein Veri Bankası, protein yapılarını arşivleyen bir kaynak olarak kuruldu. Serbest erişilebilir olan bu veri bankası, bir protein yapısını biyolojik bir soruyu incelemek için bilmesi gereken herkes için güvenilir bir araç haline geldi.
Protein Veri Bankası açıldığında, yedi proteinin yapısını içeriyordu. Neredeyse 50 yıl sonra Google DeepMind AlphaFold2’yi eğitmek için kullandığında ise, her biri yapı biyologları olarak bilinen biliminsanları tarafından zahmetle çözümlenmiş 140.000’den fazla yapıyı barındırıyordu.
Deneycilerin ıstırabı
1970’lerin ortalarından başlayarak, Janet Thornton her birkaç ayda bir postayla güvenilir bir şekilde bir paket alıyordu. İçinde Protein Veri Bankası’na yatırılmış yeni protein yapıları hakkında veri içeren 12 inçlik bir manyetik bant vardı. Oxford Üniversitesi’nde biyofizikçi olan Thornton, yeni yapıları keşfedildikleri anda analiz edebilmek için paketi hevesle açardı. Thornton’un aldığı ilk bantta yalnızca 20 yapı vardı.
Bu proteinlerin her biri yıllarca çalışmayı temsil ediyordu. Genellikle bir doktora öğrencisi, lisansüstü okulda dört veya daha fazla yılını tek bir proteini kristalleştirmek, ondan veri toplamak ve katlanmış yapıyı anlamak için bu verileri yorumlamakla geçirirdi.
Oxford’un biyofizik bölümü, o zamanlar, dünyanın X-ışını kristalografisi merkezlerinden biriydi. Orada, 1965’te, protein kristalografisinin öncülerinden biri olan David Phillips, ilk olarak bağışıklık sisteminin bakterilere saldırmak için kullandığı bir enzimin, lizozimin yapısını belirledi. Oxford biyofizikçileri X-ışını kristalografisini kullanarak, proteinlerin elektron yoğunluğunun haritalarını oluşturdular; elektronların toplandığı alanlarda muhtemelen bir atom bulunuyordu. Thornton ve meslektaşları, bu elektron yoğunluğu haritalarını plastik levhalara bastılar ve proteinin coğrafyasının bir “kontur haritası”nı oluşturmak için bunları üst üste koydular.

Daha sonra haritaları fiziksel modellere dönüştürdüler. Plastik haritalarını, cihazı 1968’de icat eden Oxford biyofizikçisi Frederic Richards’ın adını taşıyan bir Richards kutusuna yerleştirdiler. Bir Richards kutusunun içinde, açılı bir ayna haritaları bir çalışma alanına yansıtarak araştırmacıların her bir atomun diğerlerine göre tam olarak nerede bulunduğunu görmelerini sağladı. Daha sonra modellerini top ve çubuklardan fiziksel olarak oluşturdular.
Bu yöntem zahmetli ve kısıtlayıcıydı. 1971’de, daha sonra ünlü bir kristalograf olacak olan Louise Johnson, 842 aminoasitle o zamanlar üzerinde çalışılmış en büyük protein olan fosforilazı modelliyordu. Johnson, bu modelleme için Oxford’un özellikle inşa ettiği iki katlı bir Richards kutusuna bir merdivenle tırmanmak zorundaydı.
Bir model tamamlandıktan sonra, biliminsanları protein yapısı için koordinatlar bulmak amacıyla atomlar arasındaki mesafeleri ölçmek için bir cetvel kullandılar. Bu, Thornton’un deyişiyle “arkaik” bir yöntemdi. Daha sonra koordinatları bir bilgisayara girdiler. Bilgisayarlı versiyonu, atomların bir karmaşa içinde bir araya toplandığı yoğun bir ormana benziyordu. Thornton, yapıya ancak 3 boyutlu gözlüklerle baktığında proteinin topolojisini görebildiğini söylüyor. “Çok eziyetli bir süreçti, bunun yapılmış olması inanılmaz.”
Ancak yıllar süren ıstırap dolu bu sürecin sonunda başardılar. Araştırmacılar protein yapılarına güvendiklerinde, bunu Protein Veri Bankası’na gönderdiler. 1984’e gelindiğinde 152 protein depolanmıştı. 1992’de bu sayı 747’ye çıktı.
Deneyciler fiziksel modelleriyle uğraşırken, protein biyologlarının bir başka kesimi olan hesaplamalı bilimciler farklı bir yaklaşım benimsedi. Anfinsen’in bir proteinin yapısının aminoasit dizisinden tahmin edilebilir olması gerektiği görüşünü baz alarak özgüven depolamışlardı.
Kendi kurallarını yazmak
1960’ların başında lisans öğrencisi olan John Moult fizikçi olmayı planlıyordu. Sonra protein katlama problemini duydu. Moult, “Birisi geldi ve biyolojinin biyologlara bırakılmayacak kadar önemli olduğu hakkında bir ders verdi” diyor, “kibirli bir şekilde ciddiye aldım”. Büyülenen Moult kariyerini farklı bir yöne çevirdi.
Moult mezun olduktan sonra protein kristalografisine yöneldi. Penisilini yok eden bir bakteri enzimi olan beta-laktamaz da dahil olmak üzere çeşitli proteinlerin yapılarını çözdü ve 1970’te Oxford’da moleküler biyofizik alanında doktorasını aldı. Ancak doktora sonrası çalışmalarına başladığında deneyselci yaklaşımdan sıkıldı ve büyüyen protein hesaplama alanına doğru kaymaya başladı. Deneycilerin aksine hesaplamalı biyologlar, Anfinsen’in haklı olduğunu kanıtlamak için bilgisayar algoritmaları geliştirmeye çalışıyorlardı. Amaç, bir dizi aminoasidi alıp doğru protein yapısını verecek için bir program geliştirmekti.

Biyolojik deneylerden hesaplamaya geçiş zorluydu. Moult, tek bir protein yapısını çözmeye yönelik yavaş ve dikkatli çalışmalara alışmıştı. Yeni alanında ise düzenli olarak protein katlama problemini ve ilgili alt problemleri çözdüğünü iddia eden hesaplamalı makalelerle karşılaşıyordu.
Moult kuşkuluydu. “Bu alanda yayınlanan şeyler benim alıştığım titizlikte değildi” diyor. “Bunun nedeni, bu alanda hepimizin dolandırıcı olması değil. Esas neden, ne tür hesaplamalı işleri yapıyorsanız, bunu sanal bir dünyada yapıyor olmanızdır.”
Bu sanal dünyada, doğal dünyanın kuralları işe yaramadığında hesaplamacılar kendi kurallarını yazıyorlardı. Algoritmalarını, atomların belirli bir şekilde birbirine yapışması veya proteinin her zaman sağa veya sola katlanması için tasarladılar. Zamanla, modeller gerçeklikten daha da uzaklaştı. Moult, tam kontrole sahip olduğunuz bir dünyada titizliği sürdürmenin zor olduğunu düşünse de, her iki tarafın da faydalı yanlarını görebiliyordu. Deneyciler hassas ama yavaştılar; hesaplamacılar ise hızlı ama biyofiziksel gerçekliklerden o kadar uzaktılar ki çoğu koşulda yanılıyorlardı. Her iki yaklaşımın en iyilerini bir araya getirmenin bir yolu olmalı diye düşünüyordu.
Ayaklar yere vurulmaya başlıyor
1990’ların başında, Moult ve meslektaşı Krzysztof Fidelis, alanın kaosuna disiplin getirme fikrine sahipti. Yapı Tahmininin Kritik Değerlendirmesi veya İngilizce kısaltmasıyla CASP adını verdikleri bir topluluk bilimi deneyi kurdular.
Fikir basitti. CASP’ın organizatörleri olarak Moult ve Fidelis, yapıları yakın zamanda çözülmüş ve deneyciler tarafından kendilerine sağlanmış ancak sonuçları henüz yayınlanmamış proteinler için bir aminoasit dizileri listesi yayınlayacaklardı. Daha sonra dünyanın dört bir yanındaki hesaplama grupları, istedikleri yöntemi kullanarak proteinin yapısını tahmin etmek için ellerinden geleni yapacaklardı. Bağımsız bir biliminsanları grubu, cevaplarını deneysel olarak doğrulanmış yapılarla karşılaştırarak modelleri değerlendirecekti.
Fikir tuttu. CASP kısa sürede protein katlama problemine yönelik hesaplamalı yaklaşımlar için bir deneme alanı haline geldi. Bunlar, moleküler fiziği simüle etme temelli hesaplamalı yaklaşımların hüküm sürdüğü, yani yapay zekânın yükselişinden önceki zamanlardı. Bu etkinlik, araştırmacılar için adeta düşüncelerini meslektaşlarına karşı kamuya açık bir duruşmada savunma fırsatıydı. Thornton’un belirttiğine göre, etkinliğin bir tür yarışma olması amaçlanmamıştı ama sonunda bir yarışmaya dönüştü.
Biliminsanları her iki yılda bir, Monterey, California yakınlarındaki eski bir şapel olan ve daha önceleri Metodist inziva yeri olan Asilomar konferans merkezinde toplanıyordu. Bu konferanslar sırasında, organizatörler yarışmanın sonuçlarını duyuruyor ve hesaplamacılar yöntemleri ve yaklaşımları hakkında konuşmalar yapıyordu. Moult, katılımcıları, duyduklarından hoşlanmadıkları takdirde ayaklarını ahşap zemine vurmaya teşvik ediyordu. Monterey’de “Başlangıçta, oldukça fazla vurma sesi vardı” diyor.
Thornton’ın öğrencisi olan University College London’da biyoenformatik profesörü olan David Jones, ayak vuruşlarını “neredeyse bir davul gibiydi” diye hatırlıyor. Biyologlar, konuşmalar ayrıntılara saplanırsa ayaklarını vuruyorlardı. İddialar abartılıysa yine vuruyorlardı. Konuşmacılar tekrara düşüyor veya çok fazla ayrıntıya giriyorsa yine vuruyorlardı. Ancak Jones, bunun dostane bir tepki olduğunu, art niyetli olmadığını söylüyor.

Nedeni ne olursa olsun, ayak vuruşlarının yankıları bir konuşmacının kulaklarında çınlamaya başladığında, utanç vericiydi. Jones, “Tanrıya şükür hiç başıma gelmedi” diyor. Bir yıl, meslektaşlarıyla birlikte, aminoasit dizilerinin bilinen protein yapılarına uyacak şekilde bir araya getirilerek uygun yapının arandığı bir hesaplama yaklaşımı sundular. Çok da kötü geçmemiş. Jones, gülerek, “Oldukça memnunduk. … Ondan sonra her şey yokuş aşağı gitti” diyor. “Hayır, [aslında] eğlenceliydi.”
Padua Üniversitesi’nde biyoenformatik profesörü olan Silvio Tosatto, o zamanlar çok fazla heyecan olduğunu söylüyor. “İnsanlar doğru algoritmaya sahip oldukları için milyoner olabileceklerini, bazıları ise hemen Nobel Ödülü kazanacaklarını düşünüyordu.”
Bunların hiçbiri ilk yıllarda olmadı. O dönemde CASP başvurularının nasıl olduğu sorulduğunda, Moult duraklıyor ve “rastgele” kavramının duruma uyduğunu söylüyor. Bazı yöntemler, beklenenden daha iyi performans gösterirken (örneğin, bilinen proteinlerin yapılarını karşılaştırarak bilinmeyenlerin yapılarını çıkarsayan “homoloji modellemesi” gibi), diğerleri ise tam bir zaman kaybıydı. Moult, yapı tahminlerinin çoğunun “işkence görmüş gibi görünen nesneler” olduğunu söylüyor.
Hollanda Kanser Enstitüsü ve Utrecht Üniversitesi’nden yapısal biyolog Anastassis Perrakis,
“Onların başarısız olmasını görmek hoşuma gidiyordu” diyerek alay ediyor. Kendisi aynı zamanda CASP organizatörlerine yarışmada kullanılmak üzere deneysel olarak belirlenmiş yapılar veren kişiydi. “Bu bir rekabet değil, ancak bilimde birbirimizi kızdırmayı seviyoruz.”
Bu süreçte yavaş yavaş öncüler ortaya çıkmaya başladı. 1996’da ikinci CASP tamamlandıktan sonra David Baker adında genç bir araştırmacı Jones’a havaalanına giderken taksi paylaşma önerisinde bulundu. Baker, Jones’un konuşmasını dinlemişti ve o sıralar kendi hesaplamalı modeli üzerinde çalışıyordu. O seferki CASP için hazır değildi, ancak bunun hakkında sohbet etmek istiyordu. Jones takside onun fikirlerini dinledi ve onu bir daha göreceğini hiç ummuyordu.
1998’deki bir sonraki yarışmada Baker, Rosetta algoritmasıyla kapıları ardına kadar açtı. Baker bir anda “alt edilmesi gereken adam” haline geldi. Rosetta gibi algoritmalar, aminoasit moleküllerinin atomları arasındaki etkileşimleri modelleyerek bunların nasıl katlanacağını tahmin ediyordu. Baker, “Protein yapısının gerçekten tahmin edilebileceğini gösterdik” diyor. “Ancak yararlı olmak için yeterince iyi veya doğru değildi.”
2008’de insanlar hâlâ bilgisayarlara üstün gelebiliyordu. O sıralarda Washington Üniversitesi’nde kendi laboratuvarını yöneten Baker, oyuncuların aminoasit dizilerini protein yapılarına katladığı Foldit adlı ücretsiz bir çevrimiçi bilgisayar oyunu yarattı. Ekibi, Nature’da yayınladıkları bir makalede, insan Foldit oyuncularının protein modellemede Rosetta’dan daha iyi performans gösterdiğini bildirdi.
Ancak insan liderliği uzun sürmedi. 2010’ların başlarında, eş-evrim olarak bilinen bir yaklaşımda önemli atılımlar alanı ileriye taşıdı ve daha sonra yapay zekâ için kritik hale geldi. Onlarca yıldır var olan fikir basitti: Biliminsanları yüzlerce ila binlerce proteindeki yakından ilişkili aminoasit dizilerini karşılaştırarak mutasyona uğramış aminoasitleri belirleyebilir ve daha da önemlisi, diğerleriyle uyumlu bir şekilde mutasyona uğrayıp uğramadıklarını görebilirdi. İki aminoasit birlikte değiştiyse, muhtemelen bir şekilde bağlantılıydılar. Berkeley Lab’daki yapısal biyolog Adams, “Şunu söyleyebiliyorsunuz: ‘Bu iki şey muhtemelen uzayda birbirine yakın’” diyor.
Ancak 2010’ların başına kadar, hangi aminoasitlerin temas halinde olduğuna dair bu tür tahminler kasvetliydi. Doğrulukları % 20 ile % 24 arasında değişiyordu. Daha sonra biliminsanları istatistiksel yöntemlerinin hatalara yol açtığını fark ettiler. Bazı aminoasitler temas halinde olmadıkları halde temas halindeymiş gibi görünüyordu. Daha sonra Moult, istatistikçilerin bu tür hataların onlarca yıldır farkında olduğunu öğrendi. Geriye baktığınızda, “Nasıl bu kadar aptal olabilirim?” diye düşünürdünüz.
Hesaplamalı biyologlar istatistiksel araçları hatalardan temizlediler. 2016’ya gelindiğinde, temas tahmininin doğruluğu % 47’ye fırladı. İki yıl sonra ise % 70’e ulaştı. Baker’ın algoritması bu başarı üzerine inşa edildi: 2014’te Rosetta, bir CASP değerlendiricisinin Baker’ın protein katlama sorununu tamamen çözmüş olduğunu düşünebileceği kadar doğru iki protein yapısı üretti.
Adams, eş-evrim yaklaşımının içgörüsünün “harika” olduğunu söylüyor. Makine öğreniminin yaygınlaşmasından önce “bu alanı gerçekten ileriye taşıyan büyük şeylerden biriydi”. Ancak, alanı bu kadar ileri götürebildi. Eş-evrim, karşılaştırılacak çok sayıda benzer protein gerektiriyordu ve deneyciler, hesaplamacıların ihtiyaçlarını karşılayacak kadar hızlı protein yapılarını çözemiyorlardı.
Moult, evrimsel biyolojiden bir terim kullanarak, yılların sıçramalı bir denge içinde akıp geçtiğini söyledi. Bazen sanki bir milyar yıldır hiçbir iyi fikir evrimleşmemiş gibi hissediliyordu ve sonra heyecan verici bir şey oluveriyordu.
DERİNLİĞE DALMAK
David Jones, 2016 yılında Nature dergisinde yayınlanan yeni bir makalede geleceğe dair yeni bir bakış açısı yakaladı. Londra merkezli bir yapay zekâ ekibi olan Google DeepMind’dan araştırmacılar, derin öğrenme olarak bilinen bir yöntemi kullanan algoritmalarının Go adlı eski bir masa oyununda usta bir insan rakibini nasıl yendiğini ayrıntılı olarak anlattı. Jones hayrete düşmüştü. O zamanlar “Bir şeyler oluyor” diye düşündüğünü hatırlıyordu. “Bu derin öğrenme işine gerçekten girmem gerekecek.”
Derin öğrenme, yapay zekânın insan beyninden esinlenen bir çeşididir. Beyninizde, moleküler bilgiler nöron adı verilen birbirine bağlı bir beyin hücreleri ağı üzerinden gönderilir. Nöronların, komşu nöronlar tarafından gönderilen ve alıcı nörona ya ateş edip bir sinyal yaymasını ya da ateş etmemesini söyleyen molekülleri yakalayan dendrit adı verilen küçük kolları vardır.
Brown Üniversitesi’nde bilgisayar bilimi profesörü olan Michael Littman, “Eğer o nöronda yeterli aktivite olursa, o nöron ateş edecektir” diyor. Bu süreç, bir sonraki nörona başka bir molekül dalgasının salınmasıyla sonuçlanır.

1950’lerde bilgisayar bilimcileri, “sinir ağları” yaratmak için elektronik parçaları birbirine bağlayabileceklerini fark ettiler. Sinir ağındaki her birim, araştırmacıların bir nörona benzettiği bir düğümdür: Bir nöron, diğer nöronlardan bilgi alır, sonra bir sonrakilere doğru ateş edip etmeyeceğini hesaplar. Sinir ağlarında bilgi, bir görüntüdeki köpeği tanımak gibi belirli bir sonucu üretmek için birden fazla nöron katmanı arasında yayılır.
Ne kadar çok nöron katmanınız varsa, o kadar karmaşık hesaplamalar yapabilirsiniz. Ancak erken dönem sinir ağları yalnızca iki katmandan oluşuyordu. 1990’larda bu sayı üçe çıktı ve yirmi yıl boyunca orada kaldı. Littman, “Bundan daha derin ağları güvenilir bir şekilde nasıl oluşturacağımızı çözemedik” diyor.
Jones ve Moult da dahil olmak üzere yapısal biyologlar, 1990’lardan beri protein biliminde sinir ağlarını kullanmayı denediler, ancak ağların sığlığı ve verilerin seyrekliği onları engelledi. 2010’ların başında, bilgisayar bilimcileri daha fazla katmanın düzgün bir şekilde eğitilmesine imkân sağlayacak daha iyi yapılandırılmış sinir ağları yapmayı keşfettiler. Ağlar 20, 50, 100 ve sonra binlerce katmana kadar derinleşti. Littman, “Bunu 90’larda yaptığımız versiyonundan ayırmak için insanlar buna ‘derin öğrenme’ demeye başladı” diyor. “Çünkü makine öğrenmecileri bir şeyde iyiyse, o da seksi isimler uydurmaktır.”
Derin öğrenme yapay zekâyı dönüştürdü ve fotoğraflardaki veya seslerdeki özellikleri tanımada (ve daha sonraları oyunlarda insanları yenmede) üstün olan algoritmalar ortaya çıktı. Mart 2016’da, DeepMind’ın kurucu ortağı Demis Hassabis, Seul’de yapay zekâ sistemi AlphaGo’nun eski Go oyununda bir insan dünya şampiyonunu yendiğini izlerken, lisans öğrencisiyken Foldit oynadığı zamanları hatırladığını söylüyor. Aklında şöyle bir düşünce belirdi: DeepMind araştırmacıları Go ustalarının sezgilerini taklit eden bir algoritma yazabiliyorlarsa, biyoloji hakkında hiçbir şey bilmeyen ancak proteinleri katlayabilen Foldit oyuncularının sezgilerini taklit eden bir algoritma yazamazlar mıydı?
Chicago’daki Toyota Teknoloji Enstitüsü’nde profesör olan Jinbo Xu da protein katlama problemini çözmek için derin öğrenme kullanımının potansiyelini fark edenler arasında. Bu ağların görüntü tanımada yaptıklarından ilham aldı. O zamana kadar bilgisayar bilimcileri, görüntüleri parçalara ayırmak ve aralarındaki örüntüleri belirlemek için derin öğrenme algoritmalarını programlayan evrişimsel (convolutional) ağlarla büyük başarı elde etmişti. Xu bu tekniği protein katlamasına getirdi. Uzayda hangi aminoasitlerin birbirine yakın olduğunu temsil etmek için matris adı verilen matematiksel bir nesne kullandı, ardından verileri bir görüntü olarak evrişimsel bir ağa yükledi. Algoritma, bir proteini oluşturan atomların üç boyutlu koordinatlarını tahmin etmek için bu görüntüler arasındaki örüntüleri aradı.
Xu, 2016’da bu çalışmanın ön baskısını arxiv.org’da yayınladı (daha sonra PLOS Hesaplamalı Biyoloji’de yayımlandı) ve Moult’un söylediğine göre bu alan için “oldukça etkili” oldu. İnsanlara “derin öğrenmeyle yapabileceğiniz şeyleri” gösterdi.
Çok geçmeden protein yapı grupları derin öğrenmeyle uğraşmaya başladı. AlQuraishi ve ekibi, “uçtan uca” yöntem olarak adlandırılan, yalnızca sinir ağlarıyla protein yapısını doğrudan tahmin edebilen bir yaklaşım geliştiren ilk kişilerdi; bu pek işe yaramadı. Diğerleri ise bu kadar önemli hissettiren yeni bir yaklaşıma nasıl ayak basabileceklerini düşünmeye başladılar.
Jones, “Derin öğrenmeyle tam olarak ne yapmak istediğimi bilmiyordum, ancak derin öğrenme yapmam gerektiğini fark etmiştim” diyor.
Bu alana girmek için fon başvurularında bulunmaya başladığı sıralarda Google DeepMind’dan bir e-posta aldı. Jones’a CASP yarışması hakkında sorular sordular ve yardım teklif ettiler. Jones, o sırada anladığı tek şeyin “çok fazla bilgisayar gücümüz var” demeye getirdikleriydi diyor. Onlarla tanıştıktan sonra, Google’ın daha büyük hırsları olduğu ortaya çıktı. Ancak bunları başarmak için teknoloji devinin daha fazla bilimsel beyin gücüne ihtiyacı olacaktı.
Sahada yeni bir oyuncu
2016 yılında Jones, daha sonra AlphaFold olarak bilinecek bir proje üzerinde Google DeepMind için danışman olarak çalışmaya başladığında, John Jumper Chicago Üniversitesi’nde teorik kimya alanında doktorasını tamamlıyordu.
Jumper gençliğinde bilgisayarları nasıl programlayacağını kendi başına öğrenmişti. Ayrıca fizik konusunda da yeteneği vardı. Bu yüzden üniversiteye gitme zamanı geldiğinde, her ikisi de mühendis olan anne ve babasının onun asla iş bulamayacağından endişe etmesine rağmen matematik ve fizik okumaya karar verdi.
Jumper, “Baştan sona bir ‘evrenin yasaları’ fizikçisi olacağımı düşünüyordum” diyor. “Evren hakkında doğru bir şey keşfetme fikrini her zaman sevmiştim.”
Vanderbilt Üniversitesi’nde lisans öğrencisiyken, kuark adı verilen atom altı parçacıkların garip özelliklerini incelemek için Fermi Ulusal Hızlandırıcı Laboratuvarı’ndaki araştırmacılarla bir işbirliğine katıldı. Bir gün, araştırmacılarla öğle yemeği masasında otururken, acı bir haber aldı. “Peki, üzerinde çalıştığımız bu deney ne zaman başlayacak?” Jumper onlara bu soruyu sorduğunu hatırlıyor. Profesörlerden biri muhtemelen bunu görene kadar çoktan emekli olacağını, biraz daha yaşlı olan bir diğeri ise bunu görecek kadar yaşayamayacağını söylemişti.

“Bilim alanında bundan biraz daha kısa bir zaman diliminde çalışmak istiyordum” diyor Jumper. Lisans eğitimini tamamladıktan sonra teorik yoğun madde fiziği alanında doktora programına başladı ancak kısa süre sonra bıraktı. O zamanlar proteinlerin temel simülasyonlarını oluşturan New York merkezli bir şirket olan D.E. Shaw Research’te işe girmişti. Proteinlerin nasıl hareket ettiğini ve değiştiğini anlayarak akciğer kanseri gibi çeşitli rahatsızlıkların mekanizmalarını daha iyi anlamayı umuyorlardı.
Jumper’ın çalışmalarının potansiyel önemini ilk kez kavradığı zamandı: “Bu sağlık ve insanların yaşamlarını uzatmakla ilgili”. Jumper sonraki üç yıl boyunca şirketin molekülleri daha hızlı simüle etmek için özel olarak inşa ettikleri süper bilgisayarlarda protein hareketlerini modelledi. “Bazı haftaların Salı günleri tüm doktora çalışmam boyunca yapacağımdan daha fazla simülasyon yapıyordum” diyor.
2011’de lisansüstü okula bir şans daha verdi, bu sefer Chicago Üniversitesi’nde teorik kimya okudu. Hâlâ protein yapısı ve hareketiyle ilgileniyordu. Ancak akademinin yavaş temposundan bıkmıştı. Jumper, D. E. Shaw’da kullandığı “özel bilgisayar donanımına artık erişimim yoktu” diyor. Yapay zekâyı -“o zamanlar buna istatistiksel fizik diyorduk”- gelişmiş makineler gerektiren hızlı simülasyonların seviyesine ulaşmak için kullanıp kullanamayacağını merak ediyordu. Böylece makine öğrenimi ve sinir ağlarıyla uğraşmaya başladı.
Bu süre zarfında protein katlama problemi hakkında da düşünmeye başladı. Problemin Protein Veri Bankası’nda bulunan eğitim verileriyle çözülebileceğinden şüpheleniyordu – 2012’ye gelindiğinde 76.000’den fazla protein yapısı içeriyordu. “Verilerin yeterli olduğuna inanıyordum”, ancak “fikirler yeterli değildi”. Jumper, 2017’de Google DeepMind’ın protein yapı tahmini işine girdiğine dair bir söylenti duydu. Doktorasını yeni bitirmişti ve protein katlanmasını ve dinamiklerini simüle etmek için makine öğrenimini kullanıyordu. Araştırmacı olarak işe başvurdu. “Proje hâlâ gizliydi”. Bir görüşmede protein katlanması konusunu açtığında, DeepMind ekibi konuyu değiştiriyordu.
Ekim 2017’de DeepMind’ın Londra ofisine geldi. Jones’un danışmanlık desteğiyle ekip, AlphaFold’un geliştirilmesine çoktan başlamıştı. Jones, “Duvardan fikirler sektirdiğimiz çok eğlenceli zamanlardı” diyor. “Sonunda iyi bir temel fikir ortaya çıktı ve bunun üzerine gittiler.”
Algoritmalarını eğitmek için DeepMind ekibi, Protein Veri Bankası’ndan 140.000’den fazla yapı kullandı. Bu bilgileri bir evrişimsel ağa girdiler, ancak yapay zekâ mimarisinde pek bir değişiklik yapmadılar. Jumper, bunun “standart makine öğrenimi” olduğunu söylüyor.
2018 baharında AlphaFold, CASP’ye katılmaya ve gerçek bilim insanlarıyla yarışmaya hazırdı. Jones’ göre “Bu biraz Formula 1 yarışına benziyor”. “En iyi arabayı yaptığınızı düşünüyorsunuz, ancak diğer takımların ne yaptığını bilmiyorsunuz.” Riskler yüksekti. DeepMind ekibi, anonim olarak rekabet edip etmemeleri konusunda tartışıyordu; aşağılanma riskini almak istemediler.
“Kimse başarısız olmak istemez,” dedi Jones. “Akademide bu işin bir parçasıdır; başarısız olursunuz ve başka seçeneğiniz olmadığı için devam edersiniz. Ancak tabii ki milyarlarca dolarlık bir teknoloji şirketiyseniz, bir şey yapmaya çalışıp başarısız olmanız iyi bir görüntü olmaz.”
Sonuçlarını Google DeepMind adı altında sunmaya karar verdiler. Aralık toplantısından birkaç ay önce Jones, CASP’nin organizatörlerinden haber aldı. AlphaFold’un gerçekten iyi performans gösterdiği için DeepMind ekibinin toplantıya gelmesini önermişlerdi.
Zaferleri çok büyük değildi – protein yapılarını bir sonraki en iyi takıma kıyasla yaklaşık 2,5 kat daha iyi tahmin ediyorlardı. Ancak bu başarı bir izlenim bıraktı. “İlginç bir şeyin olduğu açıktı” diyor Moult.
Algoritmayı yeniden başlatmak
Zafer DeepMind ekibine enerji vermiş olmalıydı. Ancak protein katlama sorununu çözmeye yakın olmadıklarını biliyorlardı. Hassabis onları birkaç ay önce toplamıştı. Jumper, “Bunu çözmeye mi çalışacağız yoksa çalışmayacak mıyız?” dediğini hatırlıyor. “Eğer çalışmayacaksak, ciddi ciddi büyük etki yaratabileceğimiz sorunlar bulalım”. “Gerçekten karar verdiğimiz bir an yaşadık: Bunu çözmeye mi çalışacağız?” Çizim tahtasına geri döndüler.
Fizik, kimya, biyoloji ve hesaplama alanlarındaki zengin geçmişiyle Jumper, beyin fırtınası oturumlarına özgün fikirler getirdi. Kısa süre sonra, altı kişiden 15 kişiye çıkan ekibin liderliğini üstlendi. 2019’da Google DeepMind’da staj yapan ve daha sonra yapay zekâ odaklı bir biyoteknoloji şirketi olan Atomic AI’yı kuran Raphael Townshend’a göre “çok benzersiz bir şeyler oluyordu”.
Akademide uzmanlar genellikle birbirlerinden ayrıdır ve her biri çok az işbirliğiyle bağımsız projeler yürütür. DeepMind’da istatistik, yapısal biyoloji, hesaplamalı kimya, yazılım mühendisliği ve daha birçok alandaki uzmanlar protein katlanması problemi üzerinde birlikte çalıştılar. Ayrıca arkalarında Google’ın muazzam finansal ve hesaplama kaynakları vardı. Townshend, “Doktora öğrencisi olarak aylarca yapmam gereken şeyleri tek bir günde yapıyordum” diyor.
Townshend’e göre, Londra DeepMind ofisinin yüksek enerjisi çoğunlukla Jumper kaynaklıydı. Onun için “Gerçek bir dahi diyebilirim ve ayrıca çok mütevazı bir insan” diyor 2021’de DeepMind’da staj yapan ve şu anda Princeton Üniversitesi’nde yardımcı doçent olan bilgisayar bilimci Ellen Zhong. “Ekip tarafından çok seviliyordu.”
Jumper’ın liderliğinde AlphaFold yeniden inşa edildi. DeepMind yeni bir tür dönüştürücü (transformer) mimarisi tasarladı – Townshend’in söylediğine göre bu “son beş yılda gerçekleşen her makine öğrenimi atılımını temelde güçlendiren” bir tür derin öğrenme metoduydu. Sinir ağı, verilerin daha doğru temsillerini oluşturmak için (bizim konumuzda protein evrimi ve yapısına yönelik verileri) bağlantılarının gücünü ayarlıyordu. Bu verileri bir proteinin üç boyutlu yapısını tahmin etmek için ikinci bir dönüştürücüden geçirdi. Algoritma daha sonra, revize edilmiş verilerin bir kısmıyla birlikte dönüştürücülerinden birkaç kez daha geçirerek yapıyı daha da keskinleştirdi.
Jumper, AlphaFold2 üzerinde çalışmaya ilk başladıklarında algoritmaları için “berbattı, ancak beklediğimiz kadar değil” diyor. “Belli belirsiz bir şekilde proteine benzeyen sarmallar oluşturdu”. Ancak algoritmayı daha da geliştirdikçe, tahminlerinin verimliliğinde ve doğruluğunda muazzam artışlar fark ettiler.
Jumper, “Aslında korkutucuydu” diyor. Çok iyi çalışıyorsa, bu genellikle “yanlış bir şey yapıyorsunuz” anlamına gelir. Kontrol ettiler, ancak bir sorun yoktu. Sadece çalışıyordu.
Ekip, sistemlerinin biyologlara yardımcı olup olmayacağını görmek için dahili bir deney yapmaya karar verdi. Science, Nature ve Cell gibi üst düzey dergilerde yayınlanan ve yalnızca yeni bir protein yapısını tanımlamakla kalmayıp aynı zamanda yapıdan proteinin işlevi hakkında içgörüler üreten yaklaşık 50 makale belirlediler. AlphaFold2’nin deneycilerin zahmetli yaklaşımına dayanıp dayanamayacağını görmek istediler.
Aminoasit dizilerini girdiler. AlphaFold2 tahmin motorunu çalıştırdı. Her dizi için, makalelerde sunulan deneysel yapıya yakın bir tahmin ortaya koydu. Ancak, ekibin görüşüne göre, bu yeterince doğru değildi. Yapılar, deneycilerin proteinleri hakkında öğrendikleri temel ayrıntılardan yoksundu. Jumper’ göre bu, yarışı neredeyse bitirdiğinizi zannederken ikinci bir etabın daha olduğunu keşfetmeye benziyor.
Ekip, sonraki altı ay boyunca sistemi daha da geliştirdi, küçük küçük iyileştirmeler üst üste eklendi. Protein adayları 2020 CASP yarışması için yayınlanmadan birkaç hafta önce, başka bir yararlılık testi gerçekleştirdiler. Jumper memnundu. Google DeepMind, tahminlerini 2020 baharında CASP’ye sundu. Ve sonrasında geriye yaslanıp beklediler.
Deprem
Yaz başında, Moult bir CASP değerlendiricisinden bir e-posta aldı: “Şuna bakın, oldukça etkileyici.” E-postaya Google DeepMind tarafından çözülmüş bir protein yapısı eklenmişti. Moult gerçekten etkilenmişti, ancak bunun tek seferlik olduğunu düşünüyordu. Sonra bir e-posta daha aldı ve bir tane daha. “Bu garip” diye düşünmüştü. Üç, dört, bir sürü neredeyse mükemmel protein tahmini vardı ve hepsi DeepMind’dandı. Yaz sonuna doğru, Moult “çok, çok sıra dışı bir şeyin gerçekleştiğini hızla fark ettik” diyor.
CASP değerlendiricileri her başvuruyu, tahmin edilen protein yapısını kanıtlanmış deneysel yapısıyla karşılaştırarak puanlıyor. Model ve gerçekliğin atom atom eşleştiği mükemmel puan 100’dür. Moult, 90 puanın üzerindeki her şeyin bir algoritmanın bir proteinin yapısını etkili bir şekilde çözdüğünü göstereceğine her zaman inanmıştı. AlphaFold’un yapılarının çoğu ya 90 puan ya da onun üzerindeydi. Toplantıdan birkaç ay önce Moult, Jumper’ı arayıp haberi verdi. Jumper, “Yüksek sesle küfür ettim” diye hatırlıyor. “Eşim iyi olup olmadığımı sordu.”

Aralık 2020’de, Covid-19 salgınının başlamasından bir yıldan az bir süre sonra, Jumper sanal CASP toplantısında Zoom üzerinden AlphaFold2’yi sundu. Katılımcıların geri kalanı gibi Jones da evden izledi. “Öylece kalakalmıştım… bunun gerçekleşmesini izliyordum” diyor. Sinir ağı uzmanı olmayanlar için fikirler oldukça karmaşıktı. Yine de sonuçlar açıktı. DeepMind, protein katlama probleminin yapı tahmini kısmını çözmüştü. AlphaFold2, bir proteinin yapısını aminoasit dizisinden doğru bir şekilde tahmin edebiliyordu. Jones, “Ah, en sevdiğim konu öldü” diye düşündüğünü hatırlıyor. “DeepMind filmi çekti ve bu bir son.”
Anastassis Perrakis, yıllarca yarışma için CASP’a yayınlanmamış deneysel sonuçlar sunmuştu. Ekibinin üzerinde uzun zamandır çalıştığı bir protein için AlphaFold2’nin sonuçlarını gördüğünde şoke oldu. AlphaFold2 bunu mükemmel bir şekilde doğru tahmin etmişti. Evde karantinada yalnızken, biliminsanları protein bilimi dünyasının sonsuza dek değiştiğini düşünmekte birleşmişlerdi. Bu alanın sakinleri yeni manzaraya bakarken akıllarında tek bir soru vardı: Şimdi ne olacak?
ŞOK VE HUŞU
Yapısal biyoloji aniden yapılandırılmamış hale geldi. İlk başlarda “çok fazla ruhsal arayış” olduğunu söyledi Silvio Tosatto, CASP’nin ilk günlerinden beri yarışmıştı. Bazı yapısal biyologlar işlerinin modası geçeceğinden korkuyordu. Diğerleri savunmaya geçti ve AlphaFold2’nin doğru olmadığını iddia etti. Bazıları onlarca yıldır bu sorunu çözmeye çalışan hesaplamalı biyologlar için buruk hatırlanacak bir dönemdi. AlQuraishi, CASP’tan sonra yazdığı bir blog yazısında, çocuğu ilk kez evden ayrılan biri gibi hissettiğini anlatan bir katılımcıdan alıntı yaptı.
Ancak bu parlak yeni araç etrafındaki tedirginliklerine rağmen, birçok biliminsanı çok mutluydu. Yapısal çalışma yapmayanlar, daha geniş çaplı deneyleri için protein yapılarını belirlemek üzere yapısal biyologlarla işbirliği yapmak zorundaydı. Şimdi, sadece birkaç düğmeye basıp yapıyı kendi başlarına elde edebiliyorlardı.
Medyada, AlphaFold2 “her şeyi değiştirecek” parlak yeni yapay zekâ atılımı olarak kendine yer buldu. Ancak biliminsanlarının AlphaFold2’nin ne yapıp ne yapamayacağını anlamaları aylar ve hatta yıllar aldı. Jumper’ın konuşmasından yaklaşık altı ay sonra, Google DeepMind sonuçlarını yayınladı ve AlphaFold2’nin temel kodunu paylaştı. Perrakis, “AlphaFold2 çıktığında, ertesi gün onu GPU sunucularımıza kurmaya çalışıyorduk” diyor. Biyologlar oynamaya başlamışlardı.
“[AlphaFold2]’nin tepe taklak çakılmasını bekliyordum,” diyor Thornton. “Ama aslında izlenimim, bunun dikkate değer derecede başarılı olduğu.”
Zamanla, AlphaFold2’nin bir tehdit olmaktan ziyade araştırmayı hızlandırmak için bir katalizör olabileceği netleşmeye başladı. Yapısal biyologları işsiz bırakmak yerine, işlerini daha iyi yapmaları için onlara yeni bir araç verdi. Walden, “Bir yapısal biyoloğu yalnızca proteinlerin yapısını çözen bir teknik uzman olarak görürseniz, o zaman evet, elbette yapısal biyologlar işsiz kalır” diyor. Ancak bu, İnsan Genomu Projesi’nin bir genin dizisini tanımlayan bir makale yayınlayamadıkları için genomikçileri gereksiz hale getirdiğini söylemek gibi olurdu.
Birçok durumda, bir yapısal biyoloğun amacı bir proteinin işlevini keşfetmektir. AlphaFold2 ile, deneyler yoluyla bir yapıyı çözmek için aylarca veya yıllarca beklemek yerine dakikalar içinde bir hipotez oluşturabilirlerdi.
“Bu, yapısal biyolojiyi iyi yönden değiştiriyor, kötü yönden değil” diyor Adams. “Bu alanı çalışmak için daha heyecan verici hale getiriyor.”
Ancak, bazı insanların hayal ettiği gibi bir anda hemen her türden yeni ilacın keşfine yol açmadı, aksine araştırmacılar kısa sürede bu aracın sınırlamaları olduğunu öğrendiler. AlphaFold2’nin tahminleri mükemmel değil. Perrakis, deneysel doğrulamaya ihtiyaç duyduklarını ancak süreci çok hızlandırdığını söylüyor. Şimdi öğrencileri yeni bir projeye başladıklarında, önce belirli bir proteinin yapısını tahmin etmek için AlphaFold2’yi kullanıyorlar. Daha sonra bunu doğrulamak için deneyler yürütüyorlar.

Perrakis, kendisinin ve diğer araştırmacıların bir dereceye kadar X-ışını kristalografisini kullanmaya devam edeceklerini düşünüyor. Ancak başlangıçtaki protein yapılarını geliştirmek için, birçoğu derin öğrenme tahminlerini, biyolojik örneklerin ani dondurulmasını ve elektronlarla bombardıman edilmesini içeren kriyo-EM gibi gelişmiş elektron mikroskobu teknikleriyle birleştirmeye başlıyor. Daha sonra proteinlerinin ne yaptığına dair ilginç sorulara geçebilirler. AlQuraishi, AlphaFold2’nin “turbo destekli” kriyo-EM kullandığını söyledi.
Bu değişim çoktan başladı. Haziran 2022’de Science dergisinin özel bir sayısı, insan nükleer gözenek kompleksinin (ökaryotik hücrelerin nükleer zarında bulunan, moleküllerin hücre çekirdeği ile sitoplazma arasında hareketini düzenleyen yapı) atoma yakın ölçekte yapısını ortaya koydu. 30 farklı proteinden oluşan bu devasa, karmaşık yapı, onlarca yıldır biyolojik bir bilinmezlikti. Biliminsanları, kriyo-EM tarafından çözülemeyen protein yapılarındaki boşlukları doldurmak için AlphaFold2 tahminlerini kullandılar. Jumper, diğer biliminsanlarının AlphaFold2’yi kullanarak biyolojik bir atılım yaptığı bu makaleyi görmek, “[AlphaFold]’un gerçekten önemli olduğunu anladığım an oldu” diyor.
Nükleer gözenek kompleksi gibi keşifler, protein biliminin son üç yılının zaman çizelgesinde önemli bir noktayı temsil ediyor. AlphaFold2, hastalıkları incelemek ve ilaç dağıtımı için yeni araçlar oluşturmak için kullanılan protein yapılarını zaten tahmin etti. İlaçları insan hücrelerine dağıtmak için bir moleküler şırınga tasarlamak üzere AlphaFold2’yi kullanan Broad Enstitüsü’nde moleküler biyolog olan Feng Zhang, “Bizim için çok faydalı oldu” diyor.
Bir proteinin yapısını bilmek ilaç geliştirilme süreci için önemli, çünkü bir proteinin geometrisine tutunup onun davranışını değiştirecek moleküllerin belirlenmesini kolaylaştırıyor. Bazı çalışmalar AlphaFold2 tahminlerinin bu alanda deneysel yapılar kadar yararlı olmadığını öne sürmüş olsa da, başkaları deney kadar işlevli olduklarını gösterdi. Yapay zekâ araçlarının ilaç keşfi üzerindeki tam etkisi hâlâ yeni yeni ortaya çıkmakta.
Bununla birlikte, bazı biyologlar, AlphaFold2’nin bilinen proteinlerin yapılarını ve işlevlerini ayırt etmedeki kullanımının ötesine bakıyor ve doğada bulunmayan proteinleri tasarlamaya yöneliyor; bu, yeni ilaçlar tasarlamak için çok önemli bir yaklaşım olabilir
Sonraki sınır
Jumper’ın 2020 CASP konferansındaki konuşmasını izledikten hemen sonra Baker, Rosetta algoritması üzerinde çalışmaya geri döndü. Google henüz AlphaFold2’nin temel kaynak kodunu paylaşmamıştı. Yine de Baker, “Onların tanıttığı fikirlerden bazılarıyla oynamaya başladık” diyor. Google DeepMind’ın AlphaFold2’yi Nature’da yayınladığı gün, kendisi ve ekibi, AlphaFold’a oldukça doğru bir rakip olan RoseTTAFold’u duyurdu. RoseTTAFold da protein yapılarını tahmin etmek için derin öğrenmeyi kullanıyor ancak AlphaFold2’den çok farklı bir temel mimariye sahip.
Tosatto, “Bilimsel bir fikir ortaya çıktığında, en azından yeterli kaynağa sahip olanların bunu tersine mühendislik yoluyla geliştirmesi ve bunun üzerine inşa etmeye çalışması mümkün” diyor.
RoseTTAFold yalnız değildi. Meta da dahil olmak üzere diğer AlphaFold rakipleri, protein yapısı tahminini veya ilgili sorunları ele almak için kendi algoritmalarını oluşturdular. Townshend’in biyoteknoloji girişimi Atomic AI da dahil olmak üzere bazıları, RNA yapılarını anlamak için derin öğrenmeyi kullanmak suretiyle proteinlerin ötesine geçtiler. Ancak, tekil yapı tahminleri alanında, şimdiye kadar hiç kimse AlphaFold’un doğruluğuna ulaşamadı diyor Thornton. “Bunu başaracaklarından eminim, ancak bunun gibi bir… AlphaFold anı daha yakalamanın çok zor olacağını düşünüyorum.”
En azından kamuoyunda, Baker ve Jumper, CASP tarafından kurulan üretken rekabet geleneğini sürdürdüler. Baker, “Benim onlarla rekabet ettiğimi düşünebilirler, ancak bana göre onlar sadece bizim için ilham kaynağı oldular” diyor. Jumper bunu memnuniyetle karşılıyor. “İnsanların bu bilimi geliştirmesi gerçekten önemli” diyor. “AlphaFold’un entelektüel bir neslinin [devamlılığının] olmaması benim için üzücü olurdu.”
Baker, protein biliminde yeni bir sınıra odaklanmak için programının neslini şimdiden geliştiriyor; yeni hedef protein tasarımı. Şu anda, biyologlar doğanın icat ettiği proteinleri incelemekle kısıtlılar. Baker, yeni proteinler tasarlayabilecekleri bir bilim öngörüyor; Güneş ışığından yararlanmak, plastiği parçalamak veya ilaç veya aşıların temelini oluşturmak için özel olarak tasarlanmış proteinler. Hollanda’daki Hubrecht Enstitüsü’nde Baker’ın yanında doktora sonrası araştırma yapan yapısal biyolog Danny Sahtoe, “Doğada şu anda farklı protein yapılarının veya şekillerinin sayısı oldukça sınırlıdır” diyor. “Teoride daha fazlası mümkün olmalı ve daha fazla şekle sahip olabiliyorsanız, bu daha fazla işleve sahip olabileceğiniz anlamına da gelir.”
Washington Üniversitesi’ndeki Protein Tasarım Enstitüsü’nü yöneten Baker, protein tasarımının esasen “ters protein katlama problemi” olduğunu söyledi. Derin öğrenme algoritmasına bir aminoasit dizisi besleyip ondan bir protein yapısı çıkarmasını istemek yerine, bir protein tasarımcısı bir yapıyı bir algoritmaya besler ve ondan bir dizi çıkarmasını ister. Daha sonra, tasarımcı bu aminoasit dizisini kullanarak laboratuvarda proteini oluşturur.
AlphaFold ve RoseTTAFold tek başlarına bu dizileri çıkaramazlar; tam tersini yapmak üzere programlanmışlardır. Ancak Baker ve ekibi, sinir ağı mimarisine dayanarak “RoseTTAFold difüzyon” (veya RF difüzyon) olarak bilinen tasarıma özgü yeni bir RoseTTAFold versiyonu geliştirdi.
Sahtoe’te göre, protein tasarımı alanı uzun zamandır var, ancak derin öğrenme bunu hızlandırmış oldu. Proteinlerin gerçekçi bilgisayar modellerini tasarlama sürecini “inanılmaz derecede hızlı” hale getirdi. Uzman protein tasarımcılarının yeni bir proteinin omurgasını oluşturması eskiden haftalar veya aylar alıyordu. Şimdi ise günler içinde, hatta bazen bir gecede yapabiliyorlar.
Baker ayrıca Foldit’i güncelledi: Oyuncular protein yapıları inşa etmenin yanı sıra artık protein de tasarlıyorlar. Bu üretken bir süreç oldu. Baker’ın laboratuvarı, oyuncu tarafından tasarlanan proteinlerden birkaçı hakkında makaleler yazdı. Dünyanın en iyi Foldit oyuncularından biri şu anda Washington Üniversitesi’nde Baker’ın meslektaşlarından biriyle çalışan bir lisansüstü öğrencisi.
“Protein katlanmasını anlıyor muyuz? Pekâlâ, yeni yapılara katlanan yeni diziler tasarlayabiliyorsak, bu protein katlanması hakkında oldukça fazla şey anladığımızı gösterir” diyor Baker. “Bir anlamda, bunu protein katlama sorununa bir çözüm olarak da görebilirsiniz.”
Güven egzersizi
AlphaFold2’nin başarısı, biyologların yapay zekâya karşı tutumlarını tartışmasız bir şekilde değiştirdi. Uzun bir süre boyunca, birçok deneysel biyolog hesaplamaya güvenmiyordu. Bazı makine öğrenme yaklaşımlarının verileri olduğundan daha iyi gösterebileceğini artık anlamışlardı. Sonra Google DeepMind, “bununla ciddi bir çalışma yapabileceğinizi açıkça gösterdi” diyor AlQuraishi. Herhangi bir şüphecilik artık şu anlama geliyor: “Peki ya AlphaFold ile?”
Evrişimli ağlarda uzman hesaplamalı biyolog Xu, “Biyologlar artık tahmin sonuçlarımıza inanıyor” diyor. “Önceleri, biyologlar tahminlerimizin güvenilir olup olmadığından daima şüphe duyardı.”
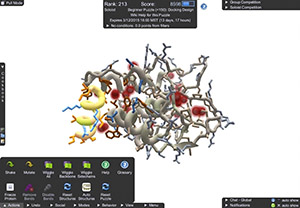
Bu güvene oynamak, AlphaFold2 platformunun bir özelliğidir: Sadece bir proteinin üç boyutlu modelini oluşturmakla kalmaz, aynı zamanda yapının farklı bölümlerini sıfırdan 100’e kadar bir güven ölçeğinde derecelendirerek tahmininin doğruluğunu kendi içinde değerlendirir.
Temmuz 2022’de, Google DeepMind 218 milyon proteinin (dünyada bilinenlerin neredeyse tamamı) yapı tahminlerini yayınladıktan sonra, Adams AlphaFold2’nin kendi kendine raporlarını analiz etmeye karar verdi. Tahminleri proteinlerin çözülmüş yapılarıyla karşılaştırdı ve doğruluklarını bağımsız olarak değerlendirdi.
Adams, “İyi haber şu ki, AlphaFold doğru tahminde bulunduğunu düşündüğünde, genellikle haklı çıkıyor” diyor. “Doğru olmadığını düşündüğünde, [tahminleri] genellikle doğru çıkmıyor”. Ancak AlphaFold2’nin tahmininde “çok emin” olduğu durumların yaklaşık % 10’unda (güven ölçeğinde en az 100 üzerinden 90 puan) yanılıyor olması gerektiğini bildirdi: Yani öngörüler deneysel olarak görülenlerle uyuşmuyordu.
Yapay zekâ sisteminin kendine bir parça şüpheyle yaklaşması, sonuçlarına aşırı güvenmeye yol açabiliyor. Çoğu biyolog, AlphaFold2’yi olduğu gibi görüyor: bir öngörü aracı. Ancak bunu çok ileri götürenler de var. Yapısal biyologlarla çalışan bazı hücre biyologları ve biyokimyacılar, onları AlphaFold2 ile değiştirdiler ve öngörülerini gerçek olarak kabul etmeye kalktılar. Perrakis, bazen biliminsanlarının, herhangi bir yapısal biyoloğa göre açıkça yanlış olan protein yapılarını içeren makaleler yayınladığını söylüyor. “Ve diyorlar ki: ‘İşte bu AlphaFold yapısı’.”
“Bazı insanlar bu derin öğrenme modellerinin neler yapabileceği konusunda aşırı özgüvenli” diyor Ulusal Sağlık Enstitüleri’nde araştırmacı olan Lauren Porter. “Bu derin öğrenme modellerini elimizden geldiğince kullanmalıyız, ancak aynı zamanda onlara dikkatli ve sağduyulu yaklaşmamız gerekiyor.”
Jones, biliminsanlarının yapıları hesaplamalı olarak belirlemek için fon bulmakta zorlandıklarını duyduğunu söylüyor. Genel algı, DeepMind’ın bunu zaten başardığı yönünde, o zaman siz neden hala uğraşasınız ki? Ancak Jones bu çalışmanın hâlâ gerekli olduğunu savunuyor çünkü AlphaFold2 yanılabilir. “Çok büyük boşluklar var” diyor Jones. “Açıkça yapamadığı şeyler var.”
AlphaFold2 küçük, basit proteinlerin yapılarını tahmin etmede mükemmel olsa da, birden fazla parça içerenleri tahmin etmede daha az doğru sonuçlar veriyor. Ayrıca proteinin çevresini veya doğada bir proteinin şeklini değiştiren diğer moleküllerle bağlarını da hesaba katamıyor. Bazen bir proteinin düzgün bir şekilde katlanması için belirli iyonlar, tuzlar veya metallerle çevrelenmesi gerekir. Walden, “Şu anda AlphaFold, bağlamı belirleyebilmekten biraz uzakta” diyor. Grubunun, AlphaFold2’nin tahmin edemediği birkaç yapıyı deneysel olarak belirlediğini de belirtelim.
Ayrıca, AlphaFold2’nin zayıf bir şekilde tahmin ettiği ancak işlev açısından daha az önemli olmayan birkaç dinamik protein türü de vardır. Şekil değiştiren proteinler (katlanma değiştiren proteinler olarak da bilinir) statik değillerdir: Şekilleri, diğer moleküllerle etkileşime girdiklerinde değişir. Bazıları, aynı aminoasit dizisine sahip olmalarına rağmen, önemli ölçüde farklı şekillerde katlanır. Katlama değiştirme proteinleri, “dizilerin tek bir yapıyı kodladığı paradigmasına meydan okuyor” diyor Porter, “çünkü açıkça öyle değiller”.
DeepMind algoritmasının eğitildiği yüz binlerce statik, tek yapılı proteinle karşılaştırıldığında, katlama değiştiren proteinlerin yalnızca yaklaşık 100 kadar örneği elimizde var. Oysa çok daha fazlasının doğada var olduğunu biliyoruz. Porter, “genel olarak konuşursak, bu algoritmaların tek bir katlamayı tahmin etmek için yapılmış olması”nın belki de şaşırtıcı olmadığını söylüyor.
Dahası bir hava dansçısı gibi çırpınan proteinler de var. İçsel olarak düzensiz proteinler veya protein bölgeleri, sabit bir yapıdan yoksundur. Sürekli olarak kıpırdanır ve yeniden form alırlar. Kopenhag Üniversitesi’nde hesaplamalı protein biyofiziği profesörü olan Kresten Lindorff-Larsen, “Birçok açıdan biraz can sıkıcı oldukları için görmezden gelindiler” diyor. İnsan proteinlerinin yaklaşık % 44’ü en az 30 aminoasitten oluşan düzensiz bir bölgeye sahiptir. Yani göz ardı edilebilecek bir olgu değil. AlphaFold2, bir bölgenin içsel olarak düzensiz olma olasılığını tahmin edebilir; ancak bu düzensizliğin neye benzediğini söyleyemez.
Jumper’ın en büyük hayal kırıklığı ise AlphaFold2’nin tek bir aminoasitle değişen iki protein arasındaki farkı, yani nokta mutasyonunu yakalayamamasıdır. Nokta mutasyonları, “bazen yapı üzerinde, sıklıkla da proteinlerin işlevi üzerinde oldukça dramatik etkilere sahip olabilir” diyor. “AlphaFold, her iki dizi için de aynı yapıyı üreteceği için onlara karşı nispeten kördür.”
Eylül 2023’te DeepMind, bu tür mutasyonların etkilerini tahmin eden bir derin öğrenme algoritması olan AlphaMissense’i yayınladı. Bu yeni program yapıdaki değişikliği gösteremese de mutasyonun proteini patojenik hale getirip getirmeyeceğini veya bilinen patojenik proteinlerdeki benzer mutasyonlara dayanarak işlev bozukluğu yaratıp yaratmayacağını kullanıcıya bildirebiliyor.
Ancak, AlphaFold2 tüm proteinleri mükemmel bir şekilde tahmin edebilse bile, biyolojik gerçekliği modellemekten hâlâ uzakta olacaktır. Bunun nedeni, bir hücrede proteinlerin asla tek başına hareket etmemesidir.
Hücresel karmaşıklık
Hücrelerin içleri karmaşık ve kaotiktir. Bir hücrenin dış zarı, yoğun bir şekilde moleküler elemanlarla dolu biyokimyasal bir ortamı sarar: proteinler, sinyal molekülleri, haberci RNA, organeller ve daha fazlası. Proteinler birbirlerine ve diğer moleküllere bağlanır, bu da formlarını ve işlevlerini değiştirir.
AlphaFold2’nin tek bir proteinin yapısını tahmin etme yeteneği, biyologları bu karmaşık doğal ortamdaki proteinleri anlamaya yaklaştırmıyor. Ancak araştırma sahası şu anda bu yöne doğru genişliyor. Protein biliminin yapay zekâ devleri, Google DeepMind ve David Baker’ın Protein Tasarım Enstitüsü, proteinlerin diğer moleküllerle etkileşime girerken aldıkları yapıları tahmin edebilmek için derin öğrenme algoritmalarını geliştiriyorlar.
2024 baharında, ikisi de bu alandaki benzer gelişmeleri anlatan makaleler yayınladı. Algoritmalarındaki güncellemeler (AlphaFold3 ve RoseTTAFold All-Atom yeni adlarıyla başlatıldı) DNA’ya, RNA’ya, diğer küçük moleküllere ve birbirlerine bağlı proteinlerin yapılarının tahmin edilebilmesini mümkün kılıyor. Biyologlar bu güncellemeleri test etmeye yeni yeni başlıyor. AlQuraishi, şimdiye kadar AlphaFold3’ün RoseTTAFold All-Atom’dan çok daha doğru tahminler ürettiğini söylüyor; bir “AlphaFold2 anı” kadar büyük bir sıçrama olmasa da. Bu programların tahmin doğruluğu, RNA yapıları gibi bazı makromoleküller söz konusu olduğunda diğer fizik tabanlı hesaplamalı yaklaşımların ve deneylerin altında kalmaya devam ediyor.
Yine de, yeni algoritmalar doğru yönde atılmış bir adım. Proteinler ve diğer moleküller arasındaki etkileşimler, hücrelerdeki işlevleri için kritik öneme sahiptir. Proteinlere bağlanabilen ve aktivitelerini istenildiği gibi değiştirebilen ilaçlar geliştirmek için araştırmacıların bu komplekslerin neye benzediğini anlamaları gerekir. Ancak Adams, her iki algoritmanın da yakın gelecekte yeni ilaçlara yol açmasının pek olası olmadığını söylüyor. “Her iki yöntem de doğruluk açısından hâlâ sınırlı, [ancak] her ikisi de elimizdekilere kıyasla önemli gelişmeler.”
DeepMind’ın yeni ürününde bir başka önemli değişiklik daha var. AlphaFold2’nin temel kodu, diğer araştırmacıların algoritmayı inceleyebilmesi ve kendi projeleri için yeniden yapabilmesi için açık kaynaklıydı. Ancak, Google, AlphaFold3’ün kaynak kodunu paylaşmak yerine şimdiye kadar onu bir ticari sır olarak korumayı tercih etti. AlQuraishi, “En azından şimdilik, hiç kimse [AlphaFold2] ile yaptıkları gibi onu çalıştırıp kullanamaz” diyor.
AlphaFold3’ün piyasaya sürülmesinden önce bile araştırmacılar, AlphaFold2’nin farklı konformasyonlardaki proteinler hakkında yararlı bilgiler sağlayıp sağlayamayacağını görmek için test ediyorlardı. Brown Üniversitesi’nde kimya ve fizik doçenti olan Brenda Rubenstein, diğer proteinleri aktive eden bir protein türü olan kinazlarla ilgileniyordu. Özellikle kansere neden olan bir kinazın mekanizmasını anlamak istiyordu, böylece ona karşı daha hassas ilaçlar geliştirebilecekti. Ekibi, Newton yasalarını kullanarak atomların üç boyutlu koordinatlarını haritalayan fizik tabanlı bir yaklaşımdan yararlanarak kinazın yapısını modelledi. Bu iş iki buçuk yıl sürdü.
“Yaklaşık bir yıl önce, şunu söyledik: Bunu daha hızlı yapabilir miyiz?”. AlphaFold2’yi yeni bir şekilde kullanmayı denediler. Algoritmaya ilgili proteinler hakkında veri sağlayarak, farklı konformasyonlardaki kinazını % 80’den fazla doğrulukla tahmin edebileceğini buldu. AlQuraishi’e göre Rubenstein’in laboratuvarı, “AlphaFold’u doğru şekilde dürterseniz, alternatif konformasyonlar çıkarmasını sağlayabileceğiniz” birkaç laboratuvardan biri. “Bu cesaret verici.”
AlQuraishi, 2040 yılına kadar derin öğrenmenin tüm bir hücreyi ve içindeki tüm yapıları ve dinamikleri simüle edebileceğini umuyor. Ancak oraya ulaşmak hem deneysel hem de hesaplamalı tarafta sıçramalar gerektirecek.
Bir yabancının görüşü
Birçok biyolog için AlphaFold2, bekledikleri atılımdı. CASP’ın amacı, protein yapısını diziden tahmin eden hesaplama araçları yaratmaktı. Yine de çoğu kişi şu soruyu sormadan edemiyor: Bu kadar çok uzman onlarca yıldır uğraşırken, nasıl oldu da alanda nispeten yeni olan birinin protein kodunu çözebilmesi mümkün oldu? Google DeepMind’ın bilgisayar ve protein bilimcilerinden oluşan ekibinin soruna getirdiği içgörüler inkâr edilemez. Aynı zamanda, protein biliminin zemini verimliydi ve derin öğrenme devrimi yaratmaya hazırdı, diyor AlQuraishi. “Bu şeyler birdenbire ortaya çıkmaz.”
CASP 2020 geldiğinde, birçok araştırmacı yapay zekâ aracılığıyla yapı tahmininde bir atılım bekliyordu. Townshend, “Her şey o yöne doğru gidiyordu” diyor. Ancak bunun milyarlarca dolarlık bir teknoloji şirketinden gelmesini beklemiyorlardı ve bunu bu kadar erken beklemiyorlardı. Bazıları AlphaFold2’nin yeni bir bilim başarısı değil, akıllı bir mühendislik olduğunu söyledi. Bazıları David Baker’ın algoritmalarının kupayı alamamasına şaşırdı. Diğerleri ise Google DeepMind’ın eşsiz kaynakları nedeniyle daha az şaşırdı.
Her yıl yaklaşık 100 laboratuvar CASP’a katılıyor ve bunlar yapay zekâ teknolojilerini benimsemeye başlamış olsalar da “muhtemelen DeepMind’ın sahip olduğu uzmanlığa veya bilgi işlem gücüne sahip değillerdi” diyor Thornton. DeepMind “temelde sınırsız bilgi işlem gücüne erişebiliyordu”.
 Ayrıca Google’ın protein bilimindeki uzmanlık eksikliğinin onları yaratıcı olarak özgürleştirmiş olabileceğini de düşünüyor. Tek bir fikirleri vardı ve harika bir sinir ağı oluşturmaya odaklandılar. Protein biyologlarının ise yükleri daha ağırdı. Yapay zekâ araçları üzerinde çalışırken, protein katlanmasında yer alan atom düzeyindeki moleküler fiziği ve kimyayı yakalamak istediler. Oysa DeepMind’ın yaklaşımı farklıydı: Dizi verilerini 3 boyutlu bir yapıya dönüştüreceğiz ve oraya nasıl ulaştığımızın bir önemi yok. Walden, DeepMind’ın diğerleri gibi protein katlama sorununu çözmeye çalışmaktan ziyade, sadece hesapsal kaba kuvvetle uzaydaki atomların son konumlarını haritalamaya odaklandıklarını düşünüyor. “Oldukça ilginç bir şekilde, muhtemelen sorunu çözdüler.”
Ayrıca Google’ın protein bilimindeki uzmanlık eksikliğinin onları yaratıcı olarak özgürleştirmiş olabileceğini de düşünüyor. Tek bir fikirleri vardı ve harika bir sinir ağı oluşturmaya odaklandılar. Protein biyologlarının ise yükleri daha ağırdı. Yapay zekâ araçları üzerinde çalışırken, protein katlanmasında yer alan atom düzeyindeki moleküler fiziği ve kimyayı yakalamak istediler. Oysa DeepMind’ın yaklaşımı farklıydı: Dizi verilerini 3 boyutlu bir yapıya dönüştüreceğiz ve oraya nasıl ulaştığımızın bir önemi yok. Walden, DeepMind’ın diğerleri gibi protein katlama sorununu çözmeye çalışmaktan ziyade, sadece hesapsal kaba kuvvetle uzaydaki atomların son konumlarını haritalamaya odaklandıklarını düşünüyor. “Oldukça ilginç bir şekilde, muhtemelen sorunu çözdüler.”
Bazı biyologlara göre bu yaklaşım protein katlama sorununu eksik bırakıyor. Yapısal biyolojinin ilk günlerinden itibaren araştırmacılar bir aminoasit dizisinin bir proteine nasıl katlandığına dair kuralları öğrenmeyi umuyorlardı. Çoğu biyolog, AlphaFold2 ile yapı tahmin sorununun çözüldüğü konusunda hemfikir. Ancak protein katlama sorunu esasında çözülmedi. Zhong, “Şu anda, size katlanmış durumları bir şekilde söyleyebilen, ancak oraya nasıl ulaştığınızı gerçekten söyleyemeyen bir kara kutunuz var” diyor. Brown Üniversitesi’nden bilgisayar bilimcisi Littman’a göre ise sorun “bir biliminsanının çözeceği şekilde çözülmedi”.
AlphaFold2, yüz binlerce protein yapısının analizine dayanarak belirli bir aminoasit dizisinin nasıl katlanabileceğine dair kalıpları tanıyabilir. Ancak biliminsanlarına protein katlama süreci hakkında hiçbir şey söyleyemez. Johns Hopkins’te emekli biyofizik profesörü olan George Rose, “Birçok insan için bilmenize gerek yok. Umursamıyorlar.” diyor. “Ancak bilim, en azından son 500 yıldır şeylerin meydana geldiği süreci anlamaya çalışmakla meşgul.” Rose, protein tabanlı yaşamın dinamiklerini, mekanizmalarını, işlevlerini ve doğasını anlamak için, derin öğrenme algoritmalarının bize anlatamadığı “hikâyenin bütününe” ihtiyacımız olduğunu savunuyor.
Moult’a göre, makinenin anlamadığı bir şey yapması önemli değil. “Hepimiz makinelerin bizim yapamadığımız şeyleri yapmasına alışkınız. Biliyorsunuz, arabam kadar hızlı koşamam.” diyor. Bir proteini incelemeye çalışan ve sadece nasıl göründüğünü kabaca bilmesi gereken moleküler biyologlar için, oraya nasıl ulaştıkları gerçekten önemli değil. Ancak, “Gerçekten nasıl çalıştığını bilene kadar, asla % 100 güvenilir bir tahminciye sahip olamayacağız” diyor Porter. “En bilgi verici tahminleri yapabilmek için temel fiziği anlamamız gerekiyor.” AlQuraishi’e göre “Hedefi ilerletmeye devam ediyoruz”. “Bence temel sorun çözüldü, bu yüzden şimdi sıra bundan sonra ne olacağına geldi.”
Perrakis’in bazen eski iş yapma biçimlerine duyulan nostalji dalgasına kapıldığı oluyor. 2022’de ekibi, daha önceden X-ışını kristalografisi kullanarak belirledikleri mikrotübüllerin (hücrelere yapı sağlayan dev, çubuk şeklindeki moleküller) değişiminde rol oynayan bir enzimi ortaya çıkardı. “Bunu [bir daha] asla yapmayacağımı fark ettim” diyor. “Aylar süren çalışmanın ardından ilk yapının ortaya çıkması çok özel bir memnuniyetti.”
AlphaFold2 bu deneyleri geçersiz kılmadı. Aksine, ne kadar gerekli olduklarını vurguladı. Tarihsel olarak farklı iki disiplini bir araya getirerek yeni ve teşvik edici bir tartışma başlattı.
Yeni dünya
Porter’ın söylediğine göre, yetmiş yıl önce proteinlerin jelatinimsi bir madde olduğu düşünülüyordu. “Şimdi ne görebildiğimize bir bakın”: İster doğada var olsunlar ister tasarlanmış olsunlar, gözün alamayacağı kadar geniş bir protein dünyasıyla karşı karşıyayız.
Perrakis, protein biyolojisi alanının “AlphaFold’dan önce olduğundan daha heyecan verici” olduğunu görüşünde. Heyecan, yapı temelli ilaç keşfini canlandırma vaadinden, hipotezler oluşturmadaki hızlanma ve hücreler içinde gerçekleşen karmaşık etkileşimleri anlama umudundan geliyor. Çok fazla veri var ve biyologlar ister laboratuvarlarında ister bilgisayarlarının önünde olsun, tüm bunlarla ne yapacaklarını anlamaya yeni başlıyorlar. Ancak dünyanın dört bir yanında kıvılcım saçan diğer yapay zekâ atılımları gibi, bunun da bir sınırı olabilir.
AlphaFold2’nin başarısı, eğitim verilerinin kullanılabilirliğine dayanıyordu, yani sabırlı deneycilerin titiz elleriyle belirlenmiş olan yüz binlerce protein yapısından. AlphaFold3 ve ilgili algoritmalar moleküler bileşiklerin yapılarını belirlemede bir miktar başarı göstermiş olsa da, doğrulukları tek proteine odaklı öncüllerinin gerisinde kalıyor. Bunun bir nedeni de önemli ölçüde daha az eğitim verisinin mevcut olmasıdır. Thornton, protein katlama probleminin “bir yapay zekâ çözümü için neredeyse mükemmel bir örnek” olduğunu, çünkü algoritmanın tekdüze bir şekilde toplanmış yüz binlerce protein yapısı üzerinde eğitilebileceğini söylüyor. Ancak Protein Veri Bankası, biyolojide organize veri paylaşımının alışılmadık bir örneği olabilir. Algoritmaları eğitmek için yüksek kaliteli veriler olmadan, doğru tahminler yapamayacaklardır.
“Şanslıydık” diyor Jumper, “sorunla, çözülmeye hazır olduğu zamanda karşılaştık”.
Derin öğrenmenin protein katlama problemini ele almadaki başarısının diğer bilim alanlarına hatta biyolojinin diğer alanlarına taşınıp taşınmayacağını kimse bilmiyor. Ancak AlQuraishi gibi bazıları iyimser. Protein katlanmasının “aslında buzdağının sadece görünen kısmı” olduğu görüşünde. Örneğin kimyagerlerin hâlâ bilgisayar kaynağı açısından pahalı hesaplamalar yapması gerekiyor. AlQuraishi, derin öğrenmeyle bu hesaplamaların daha önce olduğundan bir milyon kat daha hızlı hesaplandığını söylüyor.
Yapay zekâ, belirli türden bilimsel soruları açıkça ilerletebilir. Ancak biliminsanlarını bilgiyi ilerletmede ancak belirli bir noktaya kadar götürebilir. AlQuraishi’ye göre “Bilim, tarihsel olarak doğayı anlamakla ilgiliydi”, yani yaşamın ve evrenin altında yatan süreçleri. Bilim, çözümler ortaya koyan ve sürece dair hiçbir bilgi içermeyen derin öğrenme araçlarıyla ilerlerse, bu gerçekten bilim midir? “Kanseri tedavi edebiliyorsanız, bunun gerçekten nasıl çalıştığını umursar mısınız?” diyor AlQuraishi. “Bu, önümüzdeki yıllarda boğuşacağımız bir soru.” Eğer birçok araştırmacı doğanın süreçlerini anlamaktan vazgeçmeye karar verirse, yapay zekâ yalnızca bilimi değil, biliminsanlarını da değiştirmiş olacaktır.
Bu arada, CASP organizatörleri farklı bir soruyla boğuşuyor: Yarışmalarına ve konferanslarına nasıl devam edecekler? AlphaFold2, CASP’nin bir ürünü ve konferansın ele almak için düzenlendiği ana sorunu çözdü. 2022’de CASP toplantısı Türkiye, Antalya’da yapıldı. Google DeepMind katılmadı, ancak ekibin varlığı hissedildi. Jones, “Az çok AlphaFold kullanan insanlardı” diyor. Bu anlamda, Google’ın yine de kazandığını söyledi.
Bazı araştırmacılar artık katılmaya daha az istekli. “Bu sonucu gördüğümde, araştırma konumu değiştirdim” diyor Xu. Diğerleri kendi algoritmalarını geliştirmeye devam ediyor. Jones hâlâ yapı tahminiyle uğraşıyor, ancak bu artık onun için daha çok bir hobi. AlQuraishi ve Baker gibi diğerleri, milyarlarca dolarlık bir şirketle rekabet etme ihtimalinden yılmadan yapı tahmini ve tasarımı için yeni algoritmalar geliştirerek yola devam ediyor.
Moult ve konferans organizatörleri zamana ayak uydurmaya çalışıyor. CASP’ın bir sonraki turu mayıs ayında katılımlara açıldı. Derin öğrenmenin RNA veya biyomoleküler kompleksler gibi yapısal biyolojinin daha fazla alanını fethetmesini umuyor. Moult, “Bu yöntem bu tek sorun üzerinde işe yaradı” diyor. “Yapısal biyolojide bununla ilişkili birçok başka sorun daha var”.
Bir sonraki toplantı Aralık 2024’te Karayip Denizi’ne karşı yapılacak. Rüzgâr dostça esiyor, tartışmalar da muhtemelen öyle olacak. Ayak sesleri çoktan dindi, en azından yüksek sesli olanlar. Bu yılki yarışmanın nasıl görüneceği herkesin tahminine kalmış. Ancak son birkaç CASP bir gösterge ise, Moult yalnızca bir şeyi beklemeyi biliyor: “Sürprizler”.
Kaynak: https://www.quantamagazine.org/how-ai-revolutionized-protein-science-but-didnt-end-it-20240626/